“基于Vegan 软件包的生态学数据排序分析
赖江山 米湘成
(中国科学院植物研究所植被与环境变化国家重点实验室,北京 100093)
摘要:群落学数据一般是多维数据,例如物种属性或环境因子的属性。多元统计分析是群落生态学常用的分析方法,排序(ordination)是多元统计最常用的方法之一。CANOCO是广泛使用的排序软件,但缺点是商业软件价格不菲,版本更新速度也很慢。近年来,R语言以其灵活、开放、易于掌握、免费等诸多优点,在生态学和生物多样性研究领域迅速赢得广大研究人员的青睐。R语言中的外在软件包“Vegan”是专门用于群落生态学分析的工具。Vegan能够提供所有基本的排序方法,同时具有生成精美排序图的功能,版本更新很快。我们认为Vegan包完全可以取代CANOCO,成为今后排序分析的首选统计工具。本文首先简述排序的原理和类型,然后介绍Vegan的基本信息和下载安装过程,最后以古田山24公顷样地内随机抽取40个20m×20m的样方为例,展示Vegan包内各种常用排序方法(PCA,RDA,CA和CCA)和排序图生成过程,希望能为R的初学者尽快熟悉并利用Vegan包进行排序分析提供参考。
> setwd("/winxp_disk2/shenzy/R/Vegan")
> gtsdata=read.table("gtsdata.txt", header=T)
> gtsenv=read.table("gtsenv.txt", header=T)
> install.packages("vegan")
Installing package(s) into ‘/home/shenzy/R/x86_64-pc-linux-gnu-library/2.15’
(as ‘lib’ is unspecified)
试开URL’http://cran.csiro.au/src/contrib/vegan_2.0-4.tar.gz'
Content type 'application/x-gzip' length 1576584 bytes (1.5 Mb)
打开了URL
==================================================
downloaded 1.5 Mb
* installing *source* package ‘vegan’ ...
** 成功将‘vegan’程序包解包并MD5和检查
** libs
gfortran -fpic -O3 -pipe -g -c cepin.f -o cepin.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c data2hill.c -o data2hill.o
gfortran -fpic -O3 -pipe -g -c decorana.f -o decorana.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c goffactor.c -o goffactor.o
gfortran -fpic -O3 -pipe -g -c monoMDS.f -o monoMDS.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c nestedness.c -o nestedness.o
gfortran -fpic -O3 -pipe -g -c ordering.f -o ordering.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c pnpoly.c -o pnpoly.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c stepacross.c -o stepacross.o
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG -fpic -O3 -pipe -g -c vegdist.c -o vegdist.o
gcc -std=gnu99 -shared -o vegan.so cepin.o data2hill.o decorana.o goffactor.o monoMDS.o nestedness.o ordering.o pnpoly.o stepacross.o vegdist.o -lgfortran -lm -lquadmath -L/usr/lib/R/lib -lR
安装至 /home/shenzy/R/x86_64-pc-linux-gnu-library/2.15/vegan/libs
** R
** data
** inst
** preparing package for lazy loading
** help
*** installing help indices
** building package indices
** installing vignettes
‘decision-vegan.Rnw’
‘diversity-vegan.Rnw’ using ‘UTF-8’
‘intro-vegan.Rnw’ using ‘UTF-8’
** testing if installed package can be loaded
* DONE (vegan)
The downloaded source packages are in
‘/tmp/RtmpmtXtEK/downloaded_packages’
> library(vegan)
载入需要的程辑包:permute
载入程辑包:‘permute’
The following object(s) are masked from ‘package:gtools’:
permute
This is vegan 2.0-4
> decorana(gtsdata)
Call:
decorana(veg = gtsdata)
Detrended correspondence analysis with 26 segments.
Rescaling of axes with 4 iterations.
DCA1 DCA2 DCA3 DCA4
Eigenvalues 0.3939 0.2239 0.09555 0.06226
Decorana values 0.5025 0.1756 0.06712 0.03877
Axis lengths 3.2595 2.5130 1.21445 1.00854
> gts.pca=rda(gtsdata)
> gts.pca
Call: rda(X = gtsdata)
Inertia Rank
Total 352.1
Unconstrained 352.1 22
Inertia is variance
Eigenvalues for unconstrained axes:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8
111.779 73.580 54.607 32.959 26.481 18.063 12.763 7.637
(Showed only 8 of all 22 unconstrained eigenvalues)
Note: 通过以上命令选择排序模型(线性模型PCA、RDA或单峰模型CA、CCA),因为Axis lengths 等同于CANOCO中的DCA分析,
DCA排序数值最大max>4选单峰,<3 选线性模型, 3<max<4 则都二者都行,这里3.2595属于此情况!总的特征根为352.1,即物种分布
的总变化量。PCA排序为每个抽所能解释的方差变化量。对于第一抽而言,111.779/352.1=31.7% 为其物种分布的解释量。
> plot(gts.pca)
>biplot(gts.pca)
因以上重叠现象严重,原因是物种分布差异打,分布不均匀的物种占据了大部分排序空间,可对物种数据进行单位方差标准化。通过scale参数实现,如下:
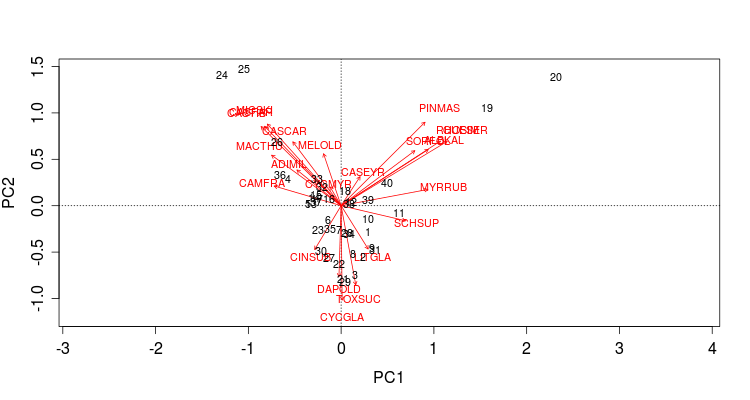
> gts.pca=rda(gtsdata, scale=T) > biplot(gts.pca,scaling=3)
Note:scaling=1 关注物种间关系
scaling=2 关注样方之间关系 scaling=3 关注样方与物种之间关系
> biplot(gts.pca,display="sp")
> biplot(gts.pca,display="si")
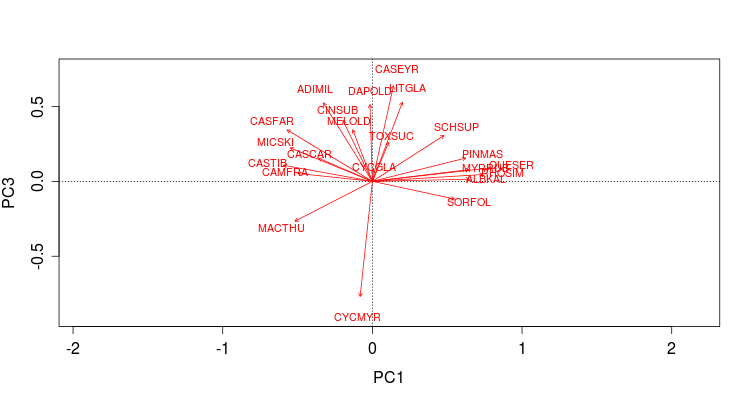
> biplot(gts.pca,display="sp", choices=c(1,3))
CA分析:
> gts.ca=cca(gtsdata)
> gts.ca
Call: cca(X = gtsdata)
Inertia Rank
Total 1.424
Unconstrained 1.424 21
Inertia is mean squared contingency coefficient
Eigenvalues for unconstrained axes:
CA1 CA2 CA3 CA4 CA5 CA6 CA7 CA8
0.50253 0.26564 0.14023 0.10502 0.09127 0.05540 0.05063 0.04204
(Showed only 8 of all 21 unconstrained eigenvalues)
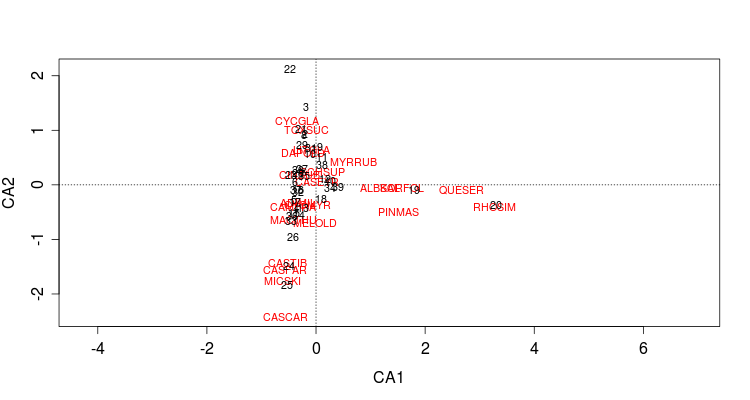
> plot(gts.ca,scaling=3)
从CA解读即:如某一个物种靠近某个样方,表明该物种可能对样方位置起很大作用。从图可以看出20号样方与短柄饱(QUESER)很近。同时19与20号样方距离近,表明物种组结构特征也近!而只有少数样方出现的物种,如CASCAR,通常在排序空间边缘,表明只偶然发生。该列对应样方数值都很小或0!对在排序中心的物种,可能在取样区域是其最优分布。对应该列(CASERY)数值较大而多!
RDA分析(多个矩阵分析):
> gts.rda=rda(gtsdata,gtsenv)
> gts.rda
Call: rda(X = gtsdata, Y = gtsenv)
Inertia Proportion Rank
Total 352.0917 1.0000
Constrained 137.4026 0.3902 8
Unconstrained 214.6891 0.6098 22
Inertia is variance
Eigenvalues for constrained axes:
RDA1 RDA2 RDA3 RDA4 RDA5 RDA6 RDA7 RDA8
56.3864 42.7769 17.8270 13.5066 2.5020 2.1217 1.6616 0.6203
Eigenvalues for unconstrained axes:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8
72.287 54.891 26.618 17.959 12.730 9.918 5.659 5.349
(Showed only 8 of all 22 unconstrained eigenvalues)
plot(gts.rda,display=c("sp","bp","si"),scaling=3)
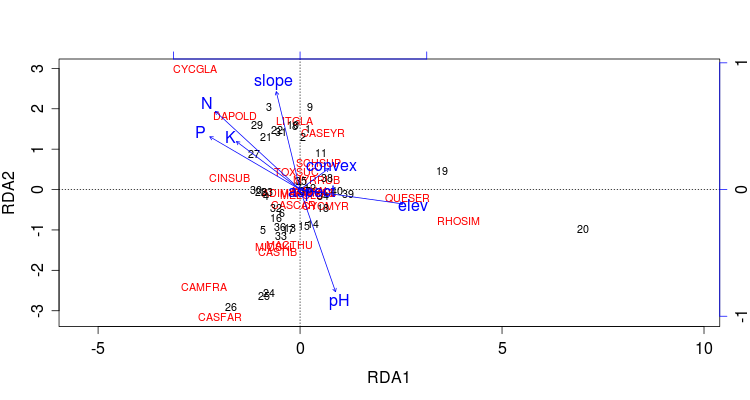
在RDA排序中,箭头连线长度代表某个
环境因子与群落分布和种类分布间相关 程度的大小,越长相关性越大。 箭头连线和排序抽的夹角代表某个环境因子 与排序抽的相关性大小,越小相关性越大!
> gts.prda=rda(gtsdata,gtsenv[,1:4], gtsenv[,5:8])
> gts.prda
Call: rda(X = gtsdata, Y = gtsenv[, 1:4], Z = gtsenv[, 5:8])
Inertia Proportion Rank
Total 352.0917 1.0000
Conditional 95.0318 0.2699 4
Constrained 42.3708 0.1203 4
Unconstrained 214.6891 0.6098 22
Inertia is variance
Eigenvalues for constrained axes:
RDA1 RDA2 RDA3 RDA4
27.522 9.087 3.442 2.320
Eigenvalues for unconstrained axes:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8
72.287 54.891 26.618 17.959 12.730 9.918 5.659 5.349
(Showed only 8 of all 22 unconstrained eigenvalues)
Note: gtsenv[,1:4]表示环境矩阵只取前4列,即地形因子。Constrained为42.37除以
352.09=12.03%,表示地形因子单独所能解释的特征根占总特征根的百分比。Y,Z调换下,
可得土壤因子单独的解释量,2者总共的解释量前面已经算出,即为39.02%。所以2组环境
变量共同的解释量为39.02%-15.53%-12.03%=11.46%!
CCA分析类似
>gts.cca=cca(gtsdata,gtsenv)

Recent Comments